Consultoría científica - Proyectos de modelización
¿Está buscando formas de simular sus datos o modelar algún proceso en particular? ¿Desea resolver un conjunto de ecuaciones diferenciales o simplemente está tratando de acelerar su algoritmo?

Entre los muchos tipos de servicios de consultoría que brindamos en XpertScientific, también desarrollamos modelos numéricos, realizamos simulaciones y podemos ayudarle a encontrar una solución eficiente para sus problemas científicos. Los científicos de XpertScientific tienen una experiencia combinada de modelado de más de 150 años y nuestras áreas de especialización incluyen biología, bioinformática, ecología, oceanografía, física, etc.
Proyectos recientes
Algunos de nuestros proyectos de modelado recientes incluyen:
- Simulaciones de Monte Carlo y Caminos Aleatorios (Random Walks) en Biología
- Trazado de rayos ópticos a través de gotas de lluvia que caen libremente
- Modelado de fotosíntesis (incluyendo fotoaclimación e inhibición) en fitoplancton
- Modelado de océanos: 3D con resolución de 1/4 de grado, utilizando batimetría realista y forzamiento externo / asimilación de datos
- Modelado de turbulencia
- Modelado de infectividad de parásitos
- Reconstrucción de filogenia, que incluye conjuntos de datos genómicos y de gran escala. Predicción de marcadores filogenéticos óptimos y códigos de barras. Datación molecular y reconstrucción del estado ancestral.
- Genómica estructural: análisis de la evolución del contenido genómico (por ejemplo, elementos transponibles, repeticiones, genes) y organización (por ejemplo, estudio de reordenamientos en regiones genómicas).
- Algoritmos de optimización / minimización de problemas (mínimos cuadrados, inversión)
Seguimiento de partículas en un estuario
La evolución de dos nubes de partículas diferentes en un estuario mediterráneo poco profundo, donde el forzamiento principal proviene del viento y del flujo de agua dulce, basada en Ross and Sharples (2004)..
Simulación cinemática (2D) de turbulencia
Simulación cinemática 2D de turbulencia basada en Fung y Vassilicos (2003). El panel izquierdo contiene la simulación cinemática donde se usan partículas de diferentes colores para visualizar la mezcla de turbulencia. El panel de la derecha muestra los histogramas correspondientes
Difracción Fraunhofer en una apertura circular
Amplitud del campo eléctrico en el detector creado por la difracción Fraunhofer en una apertura circular.
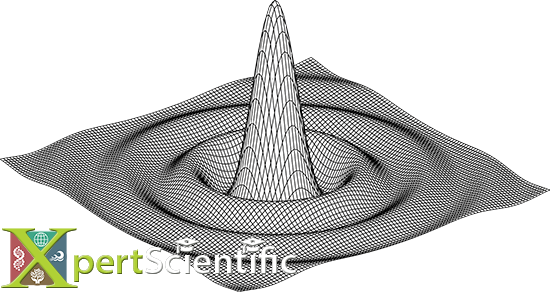
Trazado de rayos ópticos
Trazado de rayos ópticos a través de gotas de lluvia que caen libremente. El panel superior muestra el trazado del rayo a través de una gota de 3 mm, con el rayo entrante desde la izquierda, las normales de la superficie en los puntos de entrada y salida (en amarillo), los rayos reflejados (a) y doblemente refractados hacia al frente (b). El panel inferior muestra la diferencia entre las intensidades de luz polarizada “y” y “z” del patrón de refracción en el hemisferio delantero para un tamaño de gota de 3 mm, basado en un modelo Monte Carlo de Ross y Bradley (2002).
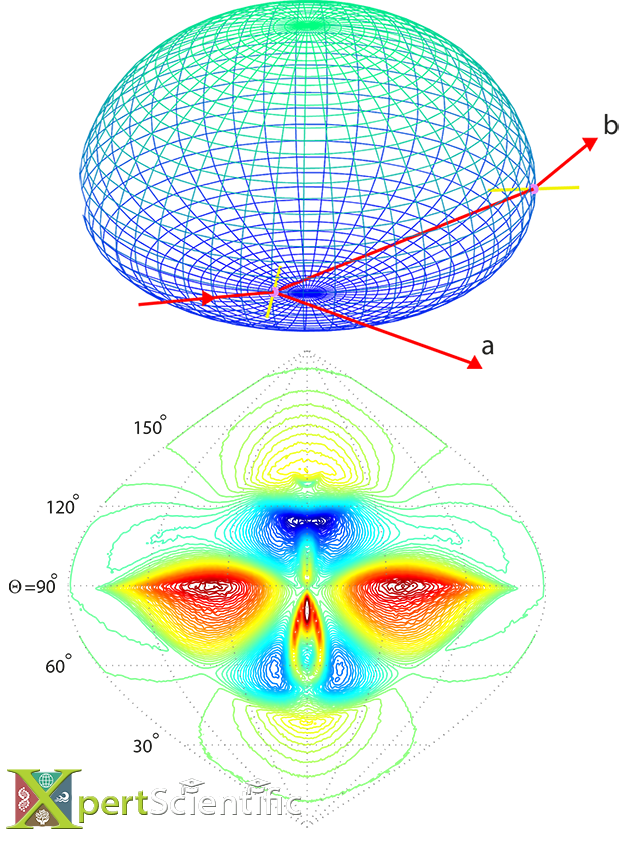
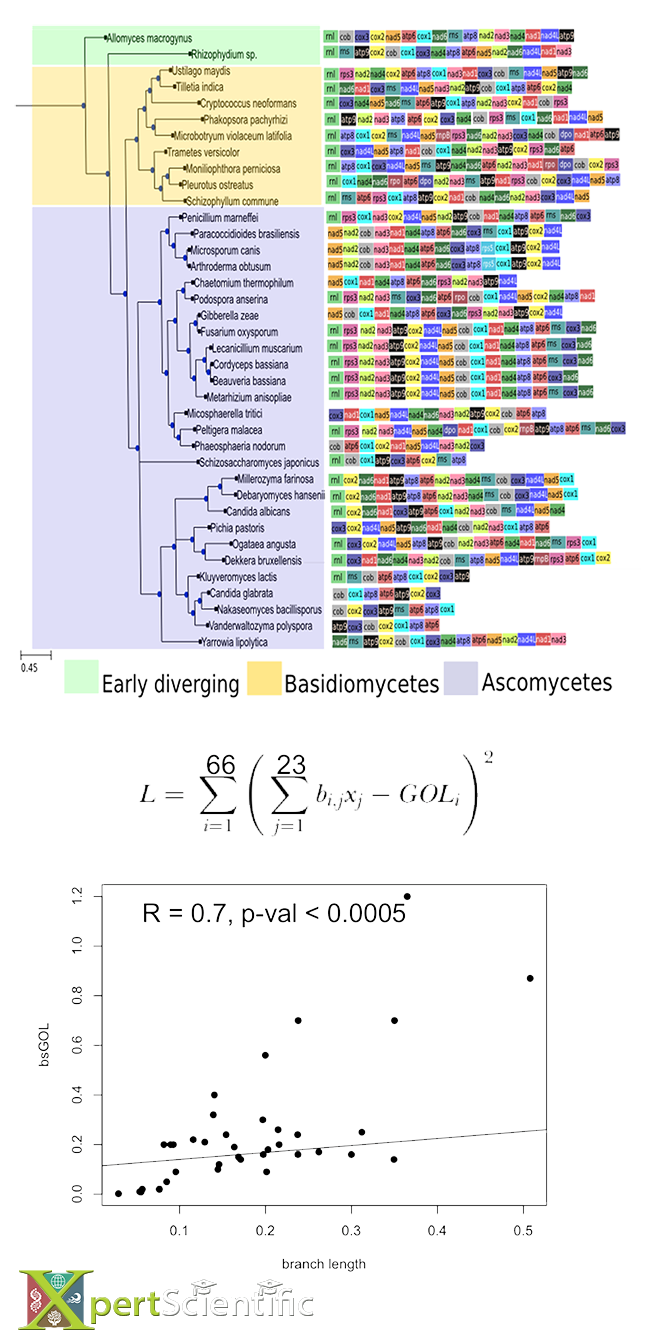
Modelado de pérdida de orden de genes
Modelado de la pérdida de orden de genes (GOL, por sus siglas en inglés) a partir de la información filogenética de genes mitocondriales en un gran grupo de hongos. El orden de genes en cada especie está representado por un conjunto de cuadros de colores. GOL se puede modelar en función de las distancias entre las especies (dadas por la longitud de las ramas), pero también por la topología del árbol.
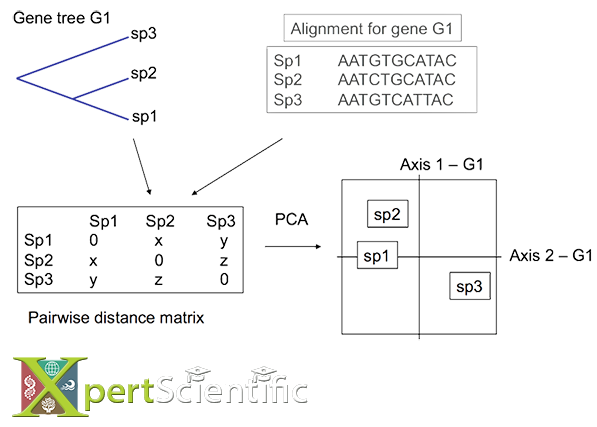
Análisis multivariable
Un enfoque gráfico multivariado para detectar especies concordantes y discordantes (anómalas) en una topología. Extraemos las distancias entre todos los pares de especies en el árbol, realizamos un análisis de componentes principales y proyectamos los ejes de separación en un solo plano para una visualización rápida.
Lenguajes de programación
Usamos los siguientes lenguajes de programación.
- C/C++
- Fortran
- Matlab
- Python
- R
Algunos de nuestros clientes