Modelisation numérique
Cherchez-vous des moyens de simuler vos données ou de modéliser un processus en particulier? Voulez-vous résoudre un ensemble d’équations différentielles ou accélérer votre algorithme?

Parmi les nombreux types de services de conseil scientifique que nous fournissons chez XpertScientific, nous développons également des modèles numériques, effectuons des simulations et pouvons vous aider à trouver une solution efficace à vos problèmes scientifiques. Les scientifiques de XpertScientific ont une expérience combinée de modélisation de plus de 150 ans et nos domaines d'expertise comprennent la biologie, la bio-informatique, l'écologie, l'océanographie, la physique, etc.
Des projets récents
Les projets récents de modélisation auxquels nous avons participé incluent :
- Simulations de Monte Carlo et Marche Aléatoire (Random Walks) en biologie
- Traçage de rayons optiques à travers des gouttes de pluie tombant librement
- Modélisation de la photosynthèse (y compris la photoacclimation et l'inhibition) dans le phytoplancton
- Modélisation de l'océan: 3D à une résolution de 1/4 degré, en utilisant une bathymétrie réaliste et forçage externe /assimilation des données
- Modèles de suivi des particules, à la fois en 1D et en 3D
- Modélisation de turbulence
- Modélisation de l'infectivité parasitaire
- Identification de gènes évoluant rapidement qui sont impliqués dans la pathogénicité fongique, la spécialisation de l'hôte et potentiellement dans la spéciation
- Développement de Phylo-MCOA: une méthode basée sur l'analyse de co-inertie multiple pour l'identification de gènes et d’espèces anomales (outliers) dans des études phylogénomiques
- Optimisation / algorithmes de minimisation des problèmes (moindres carrés, inversion)
Suivi des particules dans une baie côtière
L'évolution de deux nuages de particules dans un estuaire méditerranéen peu profond où le forçage principal provient de l'apport de vent et de l'eau douce, basée sur Ross and Sharples (2004)..
Simulation cinématique (2D) de la turbulence
Simulation cinématique 2D de la turbulence basée sur Fung et Vassilicos (2003). Le panneau à gauche montre la simulation cinématique où des particules de différentes couleurs sont utilisées pour visualiser le mélange de turbulence. Le panneau à droite montre les histogrammes correspondants.
Diffraction de Fraunhofer à une ouverture circulaire
Amplitude du champ électrique au niveau du détecteur créé par la diffraction de Fraunhofer au niveau d'une ouverture circulaire.
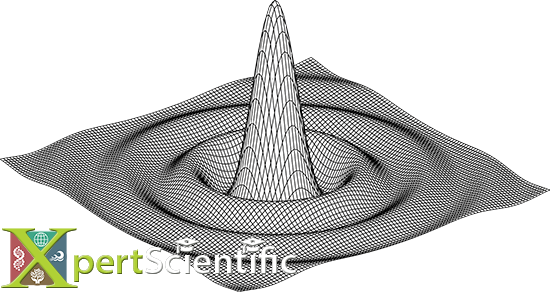
Traçage de rayons optiques
Traçage de rayons optiques à travers des gouttes de pluie tombant librement. Le panneau supérieur montre la trace d'un rayon à travers une goutte de 3mm avec le rayon entrant de la gauche, les normales de surface aux points d'entrée et de sortie (jaune), et les rayons réfléchis (a) et doublement réfractés vers l'avant (b). Le panneau inférieur montre la différence entre les intensités de lumière polarisées "y" et "z", des rayons qui ont été réfractés dans l'hémisphère avant pour une taille de goutte de 3 mm, basée sur le méthode de Monte-Carlo de Ross et Bradley (2002).
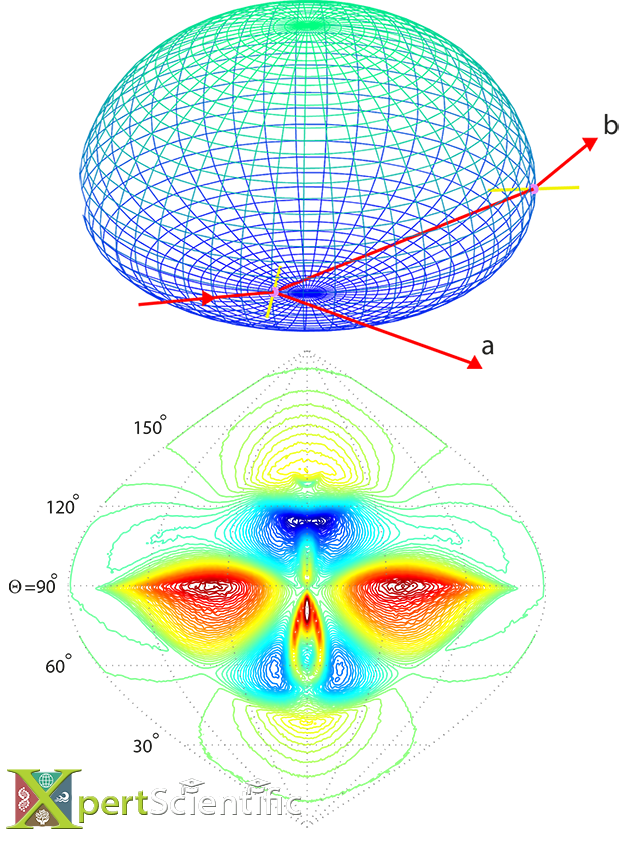
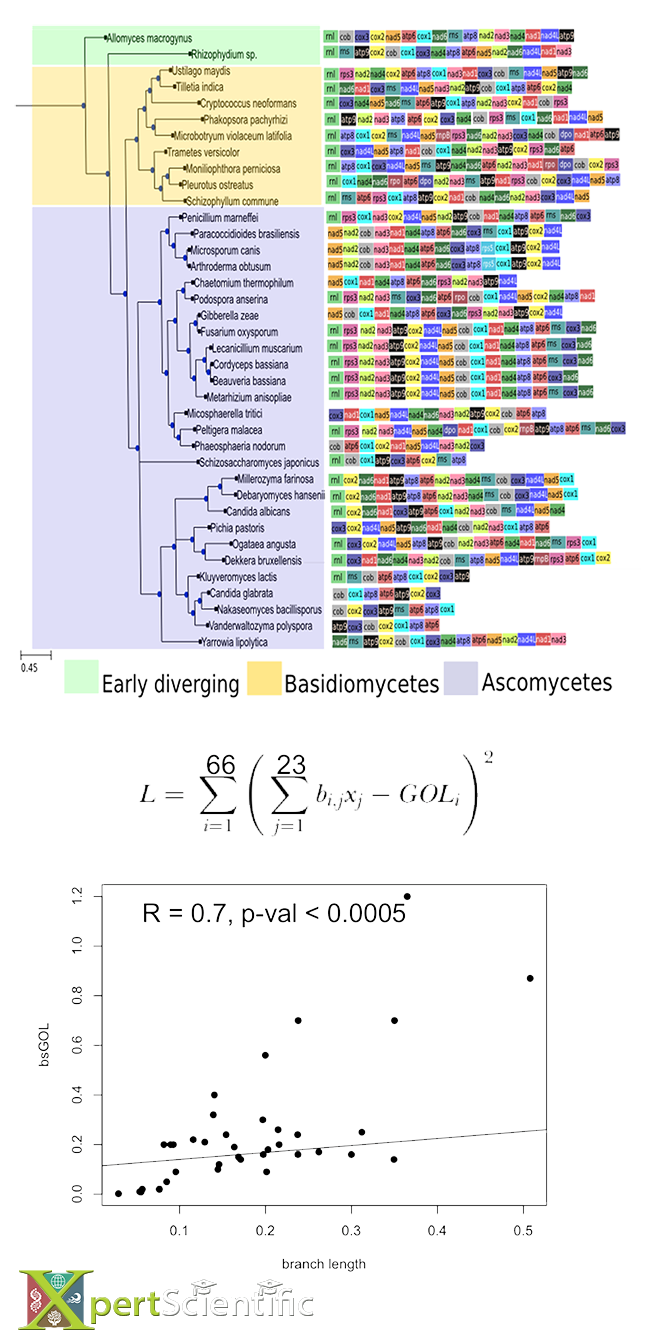
Modélisation de la perte d'ordre génétique
Modélisation de la perte d’ordre génétique (gene order loss, GOL) à partir de l’information phylogénétique des gènes mitochondriaux dans un grand groupe de champignons. L’ordre des gènes dans chaque espèce est représenté par une chaîne de cases colorées. Le GOL peut être modélisé, en fonction des distances entre espèces (données par longueurs de branches), mais aussi en utilisant la topologie de l’arbre.
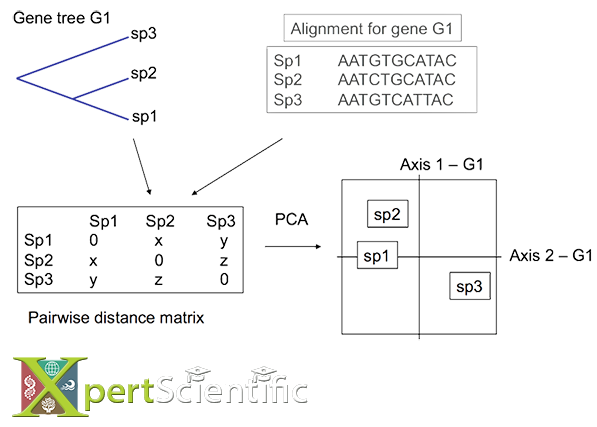
Analyse multivariée
Une approche graphique multivariée pour détecter des espèces concordantes et discordantes (aberrantes) dans une topologie. Nous calculons les distances par paires entre les espèces de l'arbre, effectuons une analyse en composantes principales et projetons les axes de séparation sur un seul plan pour une visualisation rapide.
Langages de programmation
Nous utilisons les langages de programmation suivants.
- C/C++
- Fortran
- Matlab
- Python
- R
Certains de nos clients