Numerische Modellierung und Simulationen
Suchen Sie nach Möglichkeiten, Ihre Daten zu simulieren oder einen bestimmten Prozess zu modellieren? Wollen Sie eine Reihe von Differentialgleichungen lösen oder möchten Sie lediglich Ihren Algorithmus beschleunigen?

Als Teil der zahlreichen Consulting Dienstleistungen, die wir bei XpertScientific anbieten, entwickeln wir auch numerische Modelle, führen Simulationen durch und helfen Ihnen bei der Suche nach einer effizienten Lösung für Ihre wissenschaftlichen Probleme. Die XpertScientific Wissenschaftler verfügen über eine kombinierte Modellierungserfahrung von mehr als 150 Jahren und unsere Fachgebiete umfassen Biologie, Bioinformatik, Ökologie, Ozeanographie, Physik usw.
Projekte
Einige unserer jüngsten Modellierungsprojekte umfassen:
- Monte Carlo Simulationen und Random Walks in der Biologie
- Optische Raytracing durch frei-fallende Regentropfen
- Modellierung der Photosynthese (inkl. Photoakklimatisierung & -inhibition) in Phytoplankton
- Ozeanmodellierung: 3D mit 1/4° Auflösung, mit realistischer Bathymetrie und externer Forcierung/Datenassimilierung
- Partikel Trackingmodelle, sowohl in 1D als auch 3D
- Turbulenzmodellierung
- Parasiten-Infektiositätsmodellierung
- Phylogenie-Rekonstruktion, einschließlich großer und genomischer Datensätze. Vorhersage von optimalen phylogenetischen Markern und Barcoding. Molekulare Datierung und Ahnenrekonstruktion.
- Strukturelle Genomik: Analyse der Evolution des genomischen Gehalts (z.B. transponierbare Elemente, Repeats, Gene) und Organisation (z.B. Untersuchung von Gen- und Genomregion-Umordnungen).
- Optimierungs- / Problem-Minimierungsalgorithmen (kleinste Quadrate, Inversion)
Partikel Tracking in einem Estuar
Die zeitliche Entwicklung zweier Partikelwolken in einem flachen mediterranen Estuar mit Wind-Forcing sowie Süßwassereinfluss, basierend auf der Random Walk Methode von Ross and Sharples (2004)..
Kinematische Simulation (2D) von Turbulenz
2D-Kinematische Simulation von Turbulenz, basierend auf Fung und Vassilicos (2003). Links die kinematische Simulation, bei der farbige Partikel dazu dienen die turbulente Durchmischung zu visualisieren; rechts die dazugehörigen Histogramme.
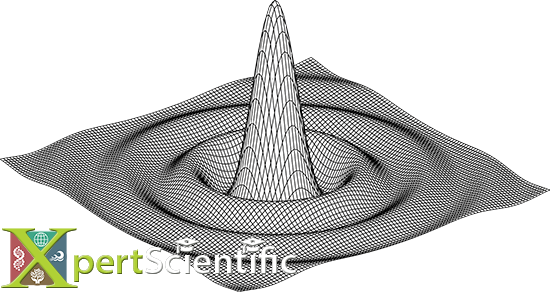
Fraunhofer-Beugung an einer kreisförmigen Öffnung
Elektrische Feldamplitude am Detektor durch Fraunhofer-Beugung an einer kreisförmigen Öffnung.
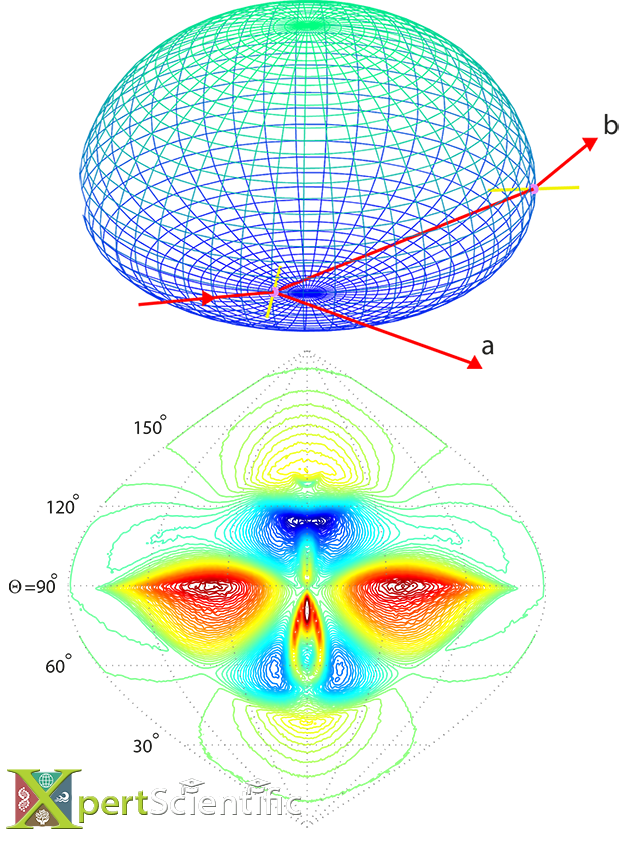
Optisches Raytracing
Optisches Raytracing durch frei fallende Regentropfen. Das obere Feld zeigt den Strahlengang durch einen 3mm-Tropfen mit dem einfallenden Lichtstrahl von links kommend, den Oberflächennormalen an den Eintritts- und Austrittspunkten (gelb) und den reflektierten (a) und den zweimal gebrochenen und vorwärts gestreuten (b) Strahlen. Das untere Bild zeigt die Differenz zwischen den y- und z-polarisierten Lichtintensitäten des Refraktionsmusters in der vorderen Hemisphäre für eine 3 mm-Tropfengröße, basierend auf dem Monte Carlo Model von Ross und Bradley (2002).
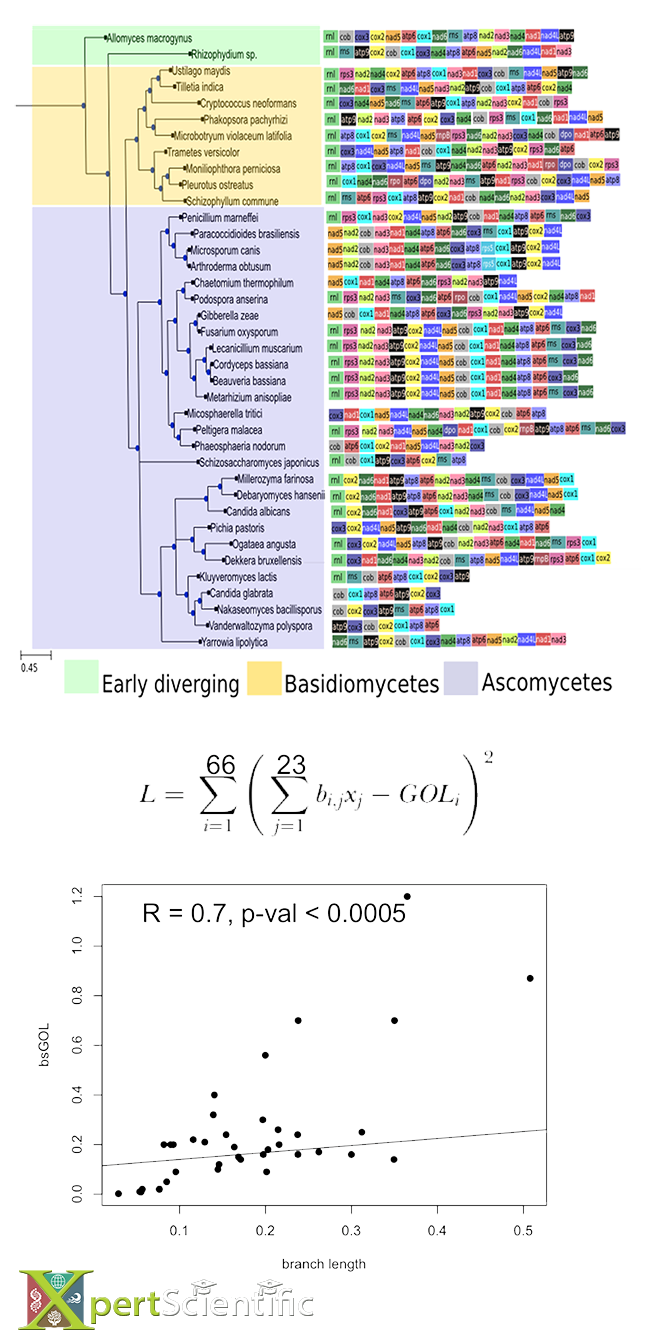
Modellierung des genetischen Ordnungsverlustes
Modellierung des Genordnungsverlustes (gene order loss, GOL) basierend auf phylogenetischen Informationen mitochondrialer Gene in einer großen Gruppe von Pilzen. Die Genordnung in jeder Spezies wird durch eine Reihe von farbigen Kästchen dargestellt. GOL kann basierend auf den Distanzen zwischen Spezies (gegeben durch Verzweigungslängen) modelliert werden, aber auch anhand der Topologie des phylogenetischen Baums.
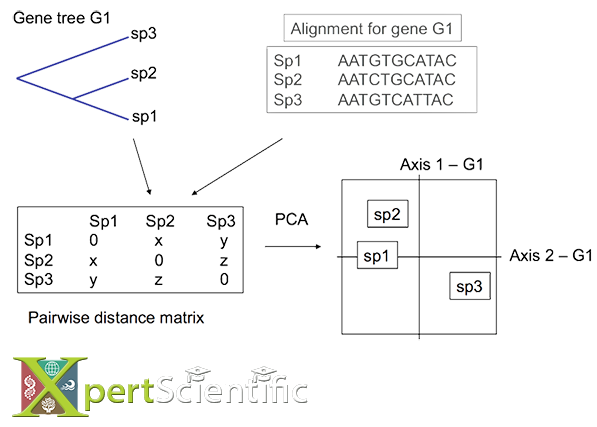
Multivariate Analyse
Ein graphischer multivariater Ansatz, um konkordante und diskordante (Ausreißer-) Spezies in einer Topologie zu erkennen. Wir extrahieren paarweise Distanzen zwischen Spezies aus dem Baum, führen eine Hauptkomponentenanalyse durch und projizieren die Separationsachsen auf eine einzige Ebene für eine schnelle Visualisierung.
Programmiersprachen
Wir verwenden die folgenden Programmiersprachen:
- C/C++
- Fortran
- Matlab
- Python
- R
Einige unserer Kunden